Prendi un algoritmo in grado di imparare attraverso l’apprendimento automatico (inDelphi). Fagli macinare una montagna di dati sperimentali sulla tecnologia più in voga nei laboratori di scienze della vita (CRISPR). Scoprirai un nuovo modo per modificare il DNA in modo prevedibile e correggere centinaia di mutazioni legate a gravi malattie genetiche. Lo ha fatto un gruppo di ricercatori di Harvard e del Massachusetts Institute of Technology, guidato da Richard Sherwood, e i risultati di questo lavoro sono stati pubblicati su “Nature”.
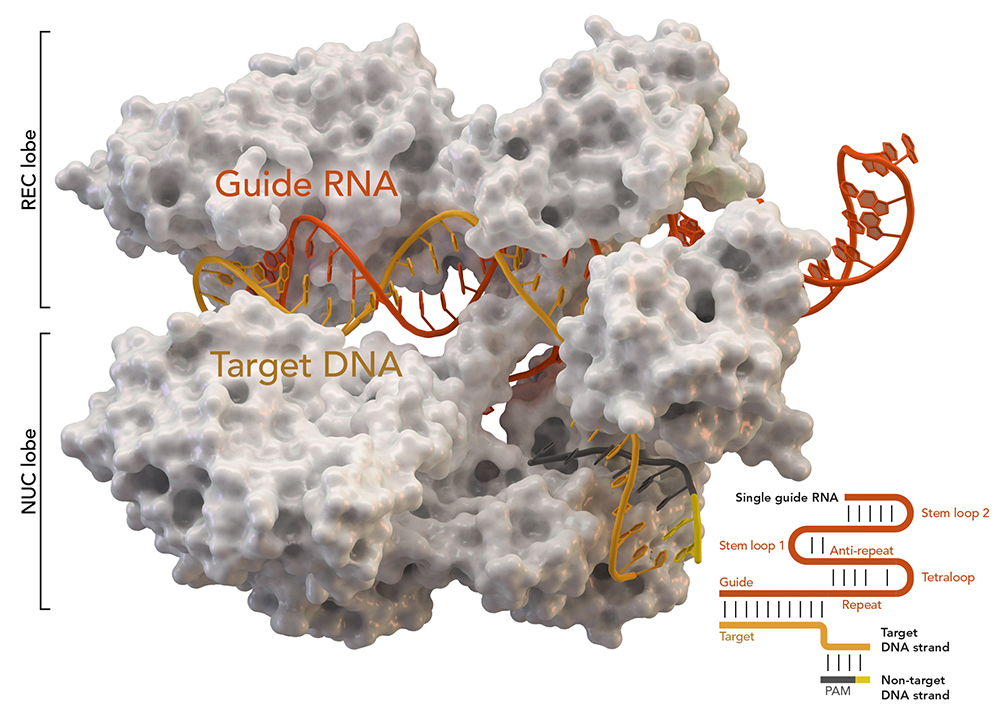
Per capire l’esperimento è utile ricordare come funziona la tecnica CRISPR. Nella versione standard si usa un enzima capace di tagliare il DNA (la nucleasi Cas9), equipaggiato con una molecola guida di RNA che funziona da bussola, identificando le sequenze da recidere con le forbici molecolari. Dopo che il DNA è stato tagliato, gli scienziati hanno sostanzialmente due opzioni di base. Possono lasciar fare ai meccanismi naturali di riparazione della cellula, senza guidare la scelta delle lettere da inserire per saldare la lesione. Oppure possono fornire uno stampo che impartisce istruzioni dettagliate.
Nel primo caso il risultato più probabile è che il gene, colpito e ricucito in modo casuale, smetta di funzionare. Nella ricerca di base, questo può essere utile per capire che funzione aveva una certa sequenza prima di essere messa ko. In campo biomedico, inoltre, il knock–out può servire per mettere a tacere un gene difettoso, evitando che continui a danneggiare l’organismo.
E ora consideriamo il secondo caso, l’opzione in cui CRISPR ricuce seguendo un copione prestabilito grazie alla presenza di uno stampo. Il risultato stavolta è una correzione mirata, che aggiusta la versione difettosa del gene facendolo funzionare a dovere. Peccato che la precisione di questo approccio abbia un prezzo in termini di efficienza, soprattutto nelle cellule che non sono attivamente in divisione.
La novità emersa grazie all’ultimo studio è che il metodo efficiente ma grossolano, quello del taglia-e-cuci senza stampo, a ben vedere non è poi tanto grossolano. Dunque, con qualche accortezza, potrebbe essere utilizzato non solo per spegnere ma anche per aggiustare i geni difettosi. Sherwood e colleghi l’hanno scoperto indirizzando CRISPR verso una varietà di siti bersaglio con 2000 molecole guida e usando l’algoritmo inDelphi e l’approccio dell’apprendimento automatico (machine learning) per prevedere il risultato delle riparazioni nel genoma umano. Ne è venuto fuori che il 5-11 per cento delle molecole guida può indurre una correzione singola e predicibile in oltre il 50 per cento dei casi.
Anche senza fornire istruzioni dettagliate a CRISPR, dunque, la modificazione genetica può risultare mirata, anziché stocastica ed eterogenea come si pensava. Per scegliere le sequenze adatte a questo tipo di correzione senza stampo (in gergo template–free editing) ci si può affidare ancora una volta a inDelphi. I ricercatori hanno già messo alla prova l’idea, correggendo efficientemente in vitro circa 200 mutazioni patogene legate a 3 malattie: sindrome di Hermansky-Pudlak, malattia di Menkes e ipercolesterolemia familiare.
“La precisione che si raggiunge è ancora troppo bassa per considerarla come un’opzione terapeutica”, commenta Anna Cereseto, che ha sviluppato una variante della Cas9 ad alta fedeltà all’Università di Trento. “Non credo che potrà sostituirsi alla ricombinazione omologa con stampo, ma il nuovo approccio sarà utile per la ricerca”, conclude la biologa molecolare.
(L’originale di questo articolo è stato pubblicato nel blog CRISPerMANIA il 7 novembre 2018. Riproduzione autorizzata, tutti i diritti riservati.)





Lascia un commento